Vol. 2° - III.6.
IL CICLO CELLULARE
Per lo più le cellule hanno vita limitata nel tempo, in quanto cessano di essere un’individualità quando si dividono in due cellule figlie dotate delle stesse caratteristiche della cellula madre.
Tutte le cellule, eccettuati i
globuli rossi e le cellule nervose, sono suscettibili di divisione, possono
cioè generare, per mitosi, due cellule figlie con le stesse caratteristiche
morfologiche e fisiologiche della cellula di partenza.
Quindi, in una cellula, si alternano mitosi e fase
intermitotica, detta anche interfase. Il ciclo cellulare è l’insieme delle
modificazioni cui va incontro una cellula da quando nasce per divisione della
cellula madre al momento in cui finisce per dividersi in due cellule figlie.
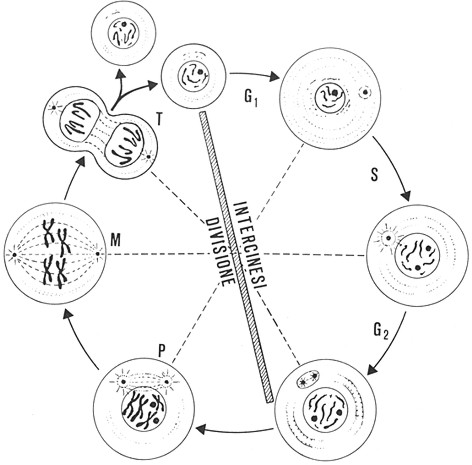
Fig. III. 24 - Il ciclo cellulare
6.1. Interfase
La maggior parte del ciclo
cellulare è occupata dall’interfase o intercinesi, compresa tra la fine di
una mitosi e l’inizio della successiva. In questo periodo il nucleo è
meccanicamente inattivo. L’interfase si suddivide in una fase G1, una fase S
e una fase G2.
6.1.a. Fase G1
La fase G1, detta anche gap
[1]
o lacuna presintetica,
segue immediatamente la mitosi. La durata di questa fase, che varia a seconda
della natura della cellula, è breve: oscilla da 5 a 10 ore in cellule di
mammiferi con elevato ritmo riproduttivo, è abbreviata nelle cellule
cancerose, talvolta è persino nulla. Ma la cellula può abbandonare il pool proliferativo per differenziarsi: allora la fase G1
corrisponderà a una fase di crescita e di differenziazione.
Processi biosintetici durante la fase G1
·
Assenza di
sintesi di DNA. Nella fase G1 non vi è sintesi di DNA: tale periodo può quindi
essere chiamato fase
di presintesi o di
preduplicazione del
DNA.
La cellula contiene la quantità di DNA caratteristica della specie, che nelle
cellule somatiche corrisponde a 2n cromosomi (2x23 nella specie umana, 2x39
nel genere Gallus). Questa quantità
rimane costante durante tutta la fase G1. In questo stadio i cromosomi sono
formati da un filamento lasso di cromatina. Nei cromocentri questo filamento
si ripiega su se stesso, mentre nelle zone in cui il nucleoplasma è chiaro i
filamenti cromosomici non sono avvolti a gomitolo, ma rimangono distesi,
costituendo così l’eucromatina.
·
Elevata
sintesi proteica. La fase G1 è la fase di crescita citoplasmatica,
caratterizzata da elevata sintesi proteica, regolata dall’attività dei
geni. Ogni gene sintetizza una molecola di RNA che ha il compito essenziale di
trasmettere al citoplasma le informazioni necessarie alla proteosintesi, da
cui il nome di RNA messaggero dato a questa molecola. Le proteine sono formate
da elementi monomerici, gli aminoacidi, la cui sequenza condiziona la
struttura specifica e le funzioni della molecola proteica. Durante la fase G1
la sintesi di RNA messaggero assicura la produzione delle proteine necessarie
alla crescita della cellula e il DNA responsabile della sintesi di tale RNA è
contenuto nell’eucromatina.
6.1.b. Fase S
La sigla S
significa sintetica.
La durata di questa fase è costante: 6¸8 ore. È caratterizzata dalla
duplicazione di tutto il DNA nucleare.
La duplicazione del DNA
Nelle cellule procariotiche la
duplicazione del DNA è
di tipo semiconservativo e quest’ipotesi appare come la più probabile.
Essa presenta le seguenti caratteristiche:
z
La replicazione ha un orientamento e inizia in un
punto particolare, detto
sito d’inizio: i filamenti parentali si separano l’uno dall’altro
nella regione dove avviene la sintesi di due nuovi filamenti, e rimangono
uniti in due punti, i punti di biforcazione o punti di crescita. La zona in
corrispondenza della quale i filamenti si presentano separati è detta occhiello
di replicazione.
z
La forca di
separazione o forca di replicazione:
a partire dai punti di crescita i due filamenti avvolti a spirale si separano,
e questo punto prende il nome di forca di separazione. Le due eliche di DNA
parentale si svolgono a una velocità che supera i 10.000 avvolgimenti/min. Il
cromosoma contiene 300.000 spire e la replicazione avviene in 30 minuti. La
despiralizzazione dipende dall’elicasi,
e la zona in cui la molecola si presenta svolta
prende il nome di occhiello di replicazione, corrispondente al tratto
di DNA compreso tra due forche di replicazione. Proteine destabilizzatrici
permettono ai due filamenti di rimanere separati in corrispondenza dell’occhiello
di replicazione.
La despiralizzazione procede in
maniera tale che l’occhiello di replicazione si allunga, provocando una
tensione sulle porzioni di DNA vicine alle forche di replicazione. La girasi nei batteri, e la topoisomerasi o proteina antielicante negli eucarioti, inducono un allentamento di
questa tensione tagliando ai due lati uno dei due filamenti.
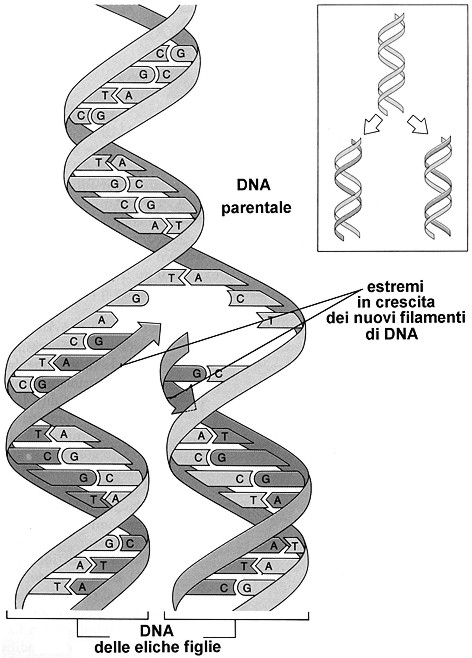
Fig. III. 25 - Duplicazione di una molecola di DNA
z
La replicazione è bidirezionale
in quanto avviene in ambedue le direzioni a partire dal sito d’inizio. Anche
nelle cellule eucariotiche
la
replicazione del DNA è semiconservativa, orientata e
bidirezionale.
Nelle cellule dei procarioti la
velocità di replicazione del DNA è dell’ordine di 2 mm/min. Se una molecola di DNA
degli eucarioti si replicasse partendo da un’estremità fino a completare il
processo all’estremità opposta, la replicazione richiederebbe 110.000
minuti, cioè 76 giorni. Invece la sua durata è di 400 minuti e ciò
significa che la replicazione avviene simultaneamente in più punti. Tali siti
sono delle unità di replicazione o repliconi, che si riproducono per conto
proprio. Negli eucarioti la loro lunghezza sarebbe pari a 40÷400 mm. Il replicone possiede un
sistema di regolazione autonomo, formato dal gene iniziatore della replicazione e dal punto d’inizio
della replicazione.
Enzimi che intervengono nella replicazione
Le DNA polimerasi isolate da Kornberg sia nel colibacillo che in
cellule eucariotiche, intervengono nella separazione dei due filamenti e nella
loro duplicazione. In vitro questi enzimi possono sintetizzare una nuova
molecola di DNA a condizione che sia presente una piccola quantità di DNA con
la funzione di stampo e a patto che siano presenti i quattro fosfonucleotidi
sotto forma di nucleotidi trifosfati. In assenza di uno solo di questi
trifosfonucleotidi la sintesi si arresta.
Nessuna DNA polimerasi nota può iniziare la sintesi di un
filamento di DNA. Le DNA polimerasi possono solo catalizzare l’addizione di
desossiribonucleotidi (dNTP) a un filamento preesistente. L’inizio della
sintesi di DNA richiede la sintesi di un corto filamento di RNA, filamento primer o innesco, catalizzata dalla DNA primasi. Vengono prodotti solamente primers
molto corti, di 3÷5 nucleotidi. I primers
funzionano come catene nucleotidiche preesistenti, cui si possono aggiungere
nuovi desossiribonucleotidi mediante reazioni catalizzate dalla DNA
polimerasi. Gli RNA primers non
faranno parte della nuova catena di DNA in quanto verranno
rimossi e rimpiazzati con DNA per azione della DNA
polimerasi.
A questo punto è necessario fare una chiara distinzione
fra primer e stampo rispetto alla replicazione del DNA:
o
il
filamento stampo o
elica stampo è quella su cui si forma la nuova elica seguendo la
legge dell’appaiamento delle basi complementari; così, la sequenza di basi
dell’elica stampo dirige la sintesi di una nuova elica con una sequenza di
basi complementare
o
il
primer è un corto segmento di nucleotidi legato all’elica
stampo, è un segmento nucleotidico che serve come substrato per l’azione
della DNA polimerasi che allunga il primer in una nuova elica di DNA, la cui
sequenza è complementare all’elica stampo.
Polarità della replicazione
Non
appena la doppia elica si apre, le DNA polimerasi assicurano la replicazione
spostandosi a leggere immediatamente il filamento principale e trascrivendo un
filamento complementare di DNA dall’estremità 3’ verso quella 5’, vale
a dire in un solo senso. Sappiamo che i due filamenti che formano una molecola
di DNA sono antiparalleli. La sintesi avviene in modo separato su ciascun
filamento: all’inizio sul filamento 1 nel senso 3’ ®
5’ per un breve tratto, poi sul filamento 2 nel senso 5’ ® 3’.
Appaiamento delle basi
La
DNA polimerasi permette alle basi complementari di appaiarsi a quelle del
filamento-stampo. Così, il filamento di DNA serve da modello, e il nuovo
filamento sintetizzato è una copia di quello che si è separato prima della
sintesi. Il filamento 1 forma un filamento
2’ o filamento
leader, copia esatta del 2, e il filamento 2 forma un filamento 1’ o filamento in ritardo,
copia esatta del filamento 1.
La lettura del filamento 2 comincia con un lieve ritardo e
porta appunto alla formazione di un filamento in ritardo. Una primasi dirige la sintesi
di un innesco di RNA complementare al filamento 2, innesco che permette a una
DNA polimerasi di sintetizzare un frammento di DNA composto da un migliaio di
nucleotidi.
Così vengono sintetizzati più frammenti di DNA, e la
sintesi di ciascun frammento avviene sempre a partire da un innesco di RNA
primer. Tali frammenti di DNA, o frammenti
di Okazaki, perdono il segmento iniziale di RNA (demolito da una
esonucleasi) dopo di che i segmenti di DNA del filamento in ritardo vengono
legati l’uno all’altro ad opera di una ligasi.
6.1.c. Fase G2
Fase
piuttosto breve, che dura 4-5 ore, nota anche come gap o lacuna
postsintetica. Inizia quando la replicazione si è completata.
Permangono attive le sintesi di RNA, e il DNA è presente in quantità doppia
rispetto alla fase G1.
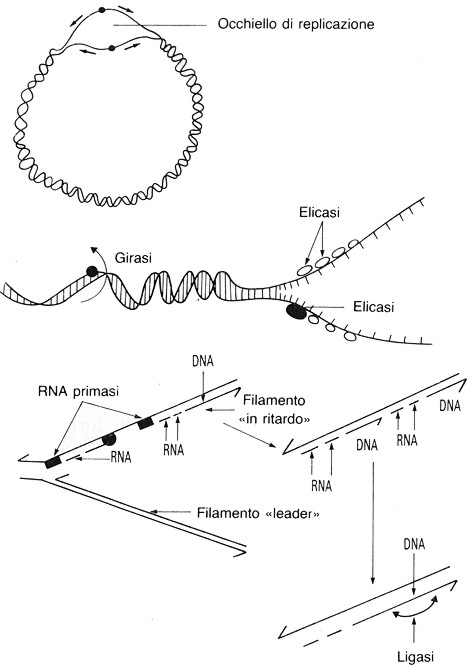
Fig.
III. 26
- Replicazione del DNA.
Il
modello è tratto dalla replicazione del DNA batterico.