Vol. 2° - I.8.
LE PROTEINE
Aminoacido
È un composto organico solubile in acqua contenente sia un gruppo carbossilico ¾ COOH, sia un gruppo aminico ¾ NH2, legati all’atomo di carbonio a.
Peptide
È un composto organico
contenente due o più aminoacidi legati tra loro da legami peptidici, formati
dalla reazione tra gruppi carbossilici e aminici adiacenti con eliminazione di
acqua. I dipeptidi contengono due aminoacidi, i tripeptidi ne contengono tre,
e così via. I polipeptidi contengono più di dieci aminoacidi, in genere 100 ¸ 300. Gli oligopeptidi con meno
di 10 aminoacidi presenti in natura sono il tripeptide glutatione e gli
octopeptidi vasopressina e ossitocina.
Proteina
È una molecola organica
costituita da una catena o da più catene lunghe di aminoacidi, cioè da
catene polipeptidiche.
Le proteine sono composti che,
per le loro peculiari caratteristiche, fanno parte di tutte le strutture
viventi e intervengono in tutte le funzioni dell’organismo.
È possibile idrolizzare completamente una proteina
mediante trattamento con acidi forti a 100°C, ottenendo così una soluzione
dei suoi monomeri costitutivi che sono gli aminoacidi. La separazione
cromatografica di questi estratti proteici ha permesso di dimostrare che sono
20 gli aminoacidi presenti in tutte le proteine naturali. L’enorme varietà
delle strutture proteiche - la cellula batterica ne possiede 102÷103
e quella eucariotica 105 - non dipende da un numero elevato di
monomeri differenti bensì dalle possibilità pressoché illimitate di
combinare i 20 diversi aminoacidi.
Una singola catena proteica può essere costituita da un
centinaio fino ad alcune migliaia di aminoacidi che possono combinarsi in
tutte le sequenze possibili, e uno stesso aminoacido può essere ripetuto più
volte, per cui è assai difficile conoscere dettagliatamente tutte le
proteine, ma, nonostante ciò, oggi è nota la struttura primaria - cioè la
sequenza aminoacidica - di oltre 4.000 proteine.
Le proteine formate esclusivamente da aminoacidi sono
dette semplici,
mentre quelle coniugate
contengono,
oltre a catene di aminoacidi che costituiscono il loro gruppo proteico, anche
molecole di natura diversa che rappresentano il gruppo
prostetico.
Sono gruppi prostetici gli ioni metallici e alcune vitamine indispensabili per
l’attività di certi enzimi, le catene oligosaccaridiche delle
glicoproteine, l’anello porfirinico di cui sono dotate l’emoglobina e
altre cromoproteine.
8.1. Gli Aminoacidi
Gli
aminoacidi sono i monomeri costitutivi delle proteine. Sono piccole molecole
organiche caratterizzate, eccetto la prolina, dalla seguente struttura comune:
|
|
COOH |
|
|
|
| |
|
|
atomo di carbonio a |
CH ¾ |
NH2 |
|
|
| |
|
|
|
R |
|
Il carbonio in posizione 2,
fatta eccezione per la glicina, è asimmetrico
in quanto legato a quattro gruppi diversi; pertanto per ogni aminoacido
possono esistere due isomeri che si designano come forme D- e L-, destrogira e levogira.
|
Aminoacidi
e loro simboli |
||
Aminoacido
|
3
lettere |
1
lettera |
|
Alanina |
Ala |
A |
|
Arginina |
Arg |
R |
|
Asparagina |
Asn |
N |
|
Acido
aspartico |
Asp |
D |
|
Asn
+ Asp |
Asx |
B |
|
Cisteina |
Cys |
C |
|
Glutamina |
Gln |
Q |
|
Acido
glutammico |
Glu |
E |
|
Gln
+ Glu |
Glx |
Z |
|
Glicina |
Gly |
G |
|
lstidina |
His |
H |
|
lsoleucina |
Ile |
I |
|
Leucina |
Leu |
L |
|
Lisina |
Lys |
K |
|
Metionina |
Met |
M |
|
Fenilalanina |
Phe |
F |
|
Prolina |
Pro |
P |
|
Serina |
Ser |
S |
|
Treonina |
Thr |
T |
|
Triptofano |
Trp |
W |
|
Tirosina |
Tyr |
Y |
|
Valina |
Val |
V |
Ciascun
aminoacido porta un gruppo chimico aggiuntivo legato al carbonio a, detto radicale
o gruppo R. È il gruppo R che varia da un
aminoacido all'altro e conferisce a ciascun aminoacido le sue proprietà
caratteristiche. Dato che le proteine hanno una sequenza e una frequenza di
aminoacidi diversa, l’organizzazione dei gruppi R conferisce alla proteina
struttura e proprietà funzionali. I 20 aminoacidi presenti in natura sono
divisi in sottogruppi, a seconda che il gruppo R sia acido (acido aspartico),
basico (lisina), neutro polare (leucina) o neutro non polare (serina).
Gli
aminoacidi di un polipeptide sono tenuti insieme da un legame peptidico, legame covalente che
unisce il gruppo carbossilico di un aminoacido al gruppo aminico dell'altro
aminoacido. Un polipeptide, quindi, è una molecola lineare, non ramificata,
che consiste normalmente 100 o più aminoacidi uniti da legami peptidici.
Ogni
polipeptide ha un gruppo a-aminico
libero a un’estremità, detta estremità
N o aminoterminale
e un gruppo a-carbossilico libero
all'altra estremità chiamata estremità
C o carbossiterminale.
I
polipeptidi hanno una polarità: per convenzione, dato che il polipeptide
viene sintetizzato secondo la direzione suddetta, l’estremità N viene definita come l’inizio della catena polipeptidica.
Nelle
proteine naturali gli aminoacidi sono sempre presenti nella forma L, mentre le
forme D non vengono utilizzate per la sintesi proteica, ma sono presenti in
alcuni piccoli polipeptidi elaborati da microrganismi, dotati di azione
antibiotica.
Gli aminoacidi sono sostanze anfotere, in quanto contengono almeno un gruppo funzionale
acido ¾ COOH e uno basico ¾
NH2. In soluzione acquosa possono essere in forma di
cationi (+), anioni (-) e anfoioni; questi ultimi sono dotati di un numero
uguale di cariche positive e negative. A pH molto bassi e molto alti
predominano le forme cationiche e anioniche. A pH intermedi è in genere
predominante la forma anfoionica.
Generalmente gli aminoacidi sono indicati con simboli a
tre lettere, ma di recente è stato proposto un sistema di simboli a una sola
lettera. Oltre agli aminoacidi costituenti le proteine, nelle cellule sono
presenti aminoacidi liberi che formano il pool
[1]
degli aminoacidi. Essi derivano dalla demolizione delle proteine o da processi
di assorbimento dal mezzo intercellulare.
8.2. Struttura delle proteine
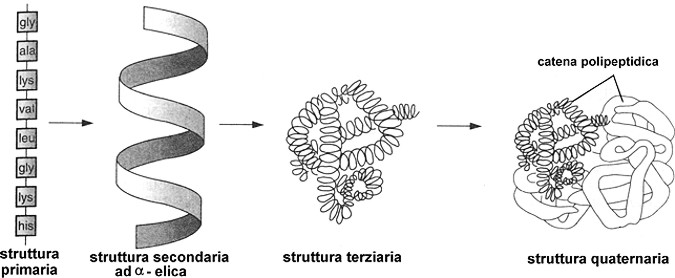
Fig. I. 5 - Livelli di organizzazione
delle proteine.
Struttura
primaria, secondaria, terziaria e quaternaria.
8.2.a. Struttura primaria delle proteine
Una proteina è formata da una
lunga catena di aminoacidi, detta sequenza
aminoacidica, in cui i monomeri sono uniti fra loro da un legame
peptidico ¾
CO ¾ NH ¾.
Tale legame, detto anche carboamidico, si forma per condensazione del gruppo
carbossilico di un aminoacido con il gruppo aminico del successivo.
La formazione del legame peptidico richiede dispendio
energetico con intervento di catalizzatori specifici, gli enzimi. I 6 atomi
costituenti il legame peptidico si trovano sullo stesso piano e ciascun legame
peptidico è unito tramite il carbonio a
al piano del legame successivo. La catena polipeptidica può quindi ripiegarsi
nello spazio solo mediante rotazioni attorno ai legami del carbonio a.
La sequenza degli aminoacidi nella catena polipeptidica
rappresenta la struttura primaria, specifica
per ogni proteina. Come si è già accennato, con 20 aminoacidi può
formarsi un numero pressoché illimitato di strutture primarie, così come con
le 21 lettere dell’alfabeto è possibile costruire un numero illimitato di
parole.
La struttura primaria, cioè tipo e sequenza degli aminoacidi, condiziona la
configurazione spaziale e la forma globale della molecola, dalle quali
dipendono le proprietà biologiche. Tuttavia l’analisi strutturale di alcune
proteine, come l’insulina, ha permesso di dimostrare che, mentre la
sostituzione anche di un solo aminoacido in certi punti della molecola porta
alla completa inattivazione funzionale della stessa, sostituzioni in altre
posizioni non hanno effetti analoghi. Ciò indica che le funzioni di una
proteina non dipendono soltanto dalla natura chimica dei suoi costituenti ma
anche dalla loro localizzazione in determinati punti della molecola,
importanti nel determinare la configurazione che la catena polipeptidica
assume nello spazio.
8.2.b. Struttura secondaria delle proteine
I legami responsabili della
formazione di una catena polipeptidica, cioè della struttura primaria, sono
sempre legami covalenti.
Come si è detto a proposito del legame peptidico,
soltanto il gruppo carboamidico è situato su di un piano fisso, mentre le
restanti parti della molecola possono avere un certo grado di libertà
rotazionale nello spazio. Ovviamente, tale grado di libertà è condizionato
dall’energia cinetica del complesso molecolare che può quindi variare
continuamente di forma, assumendo, in particolari condizioni, una struttura
elicoidale casuale, random coil.
Tuttavia, normalmente, la configurazione molecolare viene stabilizzata in un assetto
definitivo, detto appunto struttura secondaria, dall’instaurarsi
di legami deboli a idrogeno che si esercitano fino a distanze di circa 0,3 nm.
Se gruppi ¾
CO ¾
e ¾
NH ¾
appartenenti ad aminoacidi diversi e impegnati nel legame peptidico si trovano
a distanze che rientrano in tale ordine di grandezza, può stabilirsi il
legame a idrogeno che rende stabile la conformazione della catena proteica.
A causa della rigidità delle giunzioni tra legami
peptidici, è possibile soltanto un numero molto limitato di strutture
secondarie regolari, di cui le principali sono:
Ø
configurazione
tipo b-cheratina,
a fisarmonica: configurazione caratteristica di alcune molecole
proteiche a struttura allungata, dette pertanto fibrose, come le proteine
della seta e la cheratina. Tuttavia, configurazioni di questo tipo sono
possibili anche all’interno di molecole proteiche che, nell’insieme,
presentano forma globulare. Tale struttura prende origine da legami a idrogeno
tra gruppi ¾ CO ¾ e ¾ NH ¾ dei residui aminoacidici di due
catene polipeptidiche che decorrono antiparallele a distanza di circa 0,3 nm.
In tal modo si forma una struttura pieghettata a fisarmonica con i legami
carboamidici posti su di un piano e i residui aminoacidici, i gruppi laterali,
che sporgono al di sopra e al di sotto di tale lamina pieghettata.
Ø
configurazione
tipo a-elica: la struttura secondaria più
frequentemente assunta dalla catena polipeptidica è rappresentata da un
avvolgimento a spirale detto a-elica
[2]
che presenta un passo di 0,54 nm e contiene 3,6 residui aminoacidici per giro.
In questo modo la molecola polipeptidica viene ad assumere un diametro di
circa 1 nm, compresi i residui (R) che sporgono lateralmente alla spirale.
Questo tipo di struttura si forma spontaneamente se le situazioni steriche dei
residui aminoacidici lo permettono.
Ø
configurazione
tipo tropocollagene, a triplice elica: tipo particolare di
struttura secondaria presentata dalla proteina costitutiva delle fibre del
collagene, cioè il tropocollagene. Tale molecola è costituita da tre
filamenti polipeptidici (due uguali fra loro come struttura primaria e il
terzo diverso) che si organizzano in una configurazione elicoidale a passo regolare di 0,93 nm e ad andamento
sinistrorso. Ciascun filamento presenta tratti a elica regolare, dove
predominano residui aminoacidici idrofobi, alternati ad altri con struttura
più irregolare dove predominano residui idrofili. L’alta percentuale di
residui di prolina causa frequenti interruzioni nella struttura elicoidale
regolare. Quest’organizzazione strutturale può essere considerata
secondaria in quanto la maggior parte dei legami che intervengono sia nella
stabilizzazione della struttura di ogni singolo filamento che nell’associazione
ad elica regolare dei tre filamenti, sono legami a idrogeno. Tuttavia, la
struttura del tropocollagene è resa ancor più complessa dalla presenza di
radicali glucidici che intervengono nei legami laterali.
In conclusione, la struttura
secondaria di una proteina è determinata da un complesso di fenomeni sterici
che porta la molecola ad assumere una conformazione definita mediante la
formazione del massimo numero di legami a idrogeno tra gruppi ¾
CO ¾
e ¾
NH ¾
dei legami peptidici.
8.2.c. Struttura terziaria delle proteine
Dati sperimentali dimostrano che
moltissime proteine presentano una configurazione spaziale più complessa di
quella determinata dalla loro struttura secondaria. Anche tale configurazione
terziaria non è casuale ma ben definita, tanto da rendere possibile la
cristallizzazione della proteina stessa.
La struttura terziaria, che rappresenta la struttura tridimensionale vera e propria della
proteina, viene infatti determinata non solo da legami a idrogeno tra i gruppi
peptidici, ma da una serie di altri legami
di varia natura che si instaurano tra i radicali degli aminoacidi
e tra questi e il solvente, rappresentato dall’acqua, e i soluti in essa
disciolti. Tali legami sono in genere deboli, ma poiché sono presenti in gran
numero, il loro contributo totale può essere anche più stabilizzante di un
legame covalente. Tra questi tipi di interazioni, le più frequenti sono:
o
legami di tipo ione-ione e ione-dipolo che si instaurano
tra residui ionizzati di aminoacidi acidi e basici oppure tra questi residui e
l’acqua o gli ioni in essa disciolti
o
legami dipolo-dipolo (legami a idrogeno) tra gruppi
presenti nelle catene laterali (R) di alcuni aminoacidi, come tirosina e acido
aspartico
o
forze di Van der Waals dovute ad attrazioni
elettrostatiche istantanee da dipoli indotti
o
interazioni non polari che hanno luogo tra residui apolari
in base alla comune repulsione per l’acqua
o
alla stabilizzazione della struttura terziaria possono
contribuire i ponti ¾
S ¾
S ¾
che si stabiliscono fra due residui cisteinici
Finora, mediante analisi cristallografica ai raggi X, è
stata definita la struttura tridimensionale di alcune centinaia di proteine.
Come si è accennato, le proprietà chimico-fisiche e biologiche di una
proteina dipendono dall’integrità della struttura terziaria. Infatti la
funzione esplicata da una proteina è una conseguenza della specificità dei
rapporti che essa contrae con altre molecole. Questa specificità è di tipo sterico, cioè legata alla
configurazione spaziale della molecola proteica.
Nel caso degli enzimi, degli ormoni e degli anticorpi, le
reazioni chimiche mediate da proteine non dipendono tanto da interazioni
forti, come quelle che intervengono tramite legami ionici e covalenti, ma
dalle numerosissime interazioni deboli che rendono possibile il riconoscimento
di superfici molecolari complementari e richiedono quindi una localizzazione
precisa dei gruppi che debbono intervenire nella reazione. È questa infine la
ragione per cui
la
sostituzione di un singolo residuo aminoacidico può alterare completamente la
funzionalità di una proteina o, al contrario, non avere
alcun effetto. Ciò, perché, non importa tanto la natura chimica della
sostituzione, quanto il sito in cui questa avviene, potendo provocare o meno
una modificazione nella struttura tridimensionale o terziaria della
macromolecola.
8.2.d. Struttura quaternaria delle proteine
In condizioni fisiologiche
alcune proteine sono costituite da più catene polipeptidiche, dette subunità,
associate tra loro a formare un complesso proteico dotato di una definita
struttura spaziale,
la struttura quaternaria.
L’emoglobina
e la mioglobina sono tipici esempi di
proteine dotate di struttura quaternaria. Tali molecole sono costituite da
quattro catene polipeptidiche, due a due uguali, denominate a-globine e b-globine. Ogni globina ha una
caratteristica struttura terziaria che consente di accogliere, in un incavo
profondo, un gruppo tetrapirrolico contenente il ferro, detto eme, capace di
legare l’ossigeno. Tale gruppo risulta sempre unito con legami ionici alla
catena polipeptidica, mentre lo ione ferroso bivalente dell’eme è legato,
oltre che con l’azoto dei quattro nuclei pirrolici, anche con l’azoto di
un residuo istidinico.
Molte proteine presentano struttura quaternaria, essendo
costituite da monomeri proteici che, se dissociati e poi posti in condizioni
sperimentali adatte, riescono a riassociarsi in modo specifico riformando un
complesso proteico completo dotato di attività funzionale.
In conclusione, le proteine rappresentano una classe di
macromolecole altamente specifiche che svolgono funzioni essenziali quali:
§
catalisi, che consiste nel controllo
della velocità delle reazioni biochimiche
§
riconoscimento sterico,
cioè la capacità di riconoscere specifici gruppi molecolari; quest’ultima
capacità sta alla base del sistema immunitario e della trasmissione dei
messaggi ormonali, nonché del riconoscimento del substrato da parte dell’enzima
§
funzioni strutturali
nell’architettura della cellula.